Eine der größten Schwierigkeiten in der Bekämpfung von viralen Infektionskrankheiten ist die exzellente Anpassungsfähigkeit der Viren. „Bei SARS-CoV-2 haben wir gesehen, wie schnell sich ständig neue Varianten bilden, die unterschiedliche Eigenschaften mit sich bringen. Deshalb ist es wichtig, dass es uns in Zukunft gelingt, rascher und präziser vorherzusagen, wie gefährlich ein Virus werden kann“, erklärt Christian Gruber, Geschäftsführer des Bioinformatik-Start-ups Innophore, einem Spin-off der Universität Graz und des Austrian Centre of Industrial Biotechnology (acib).
Der Wissenschafter präsentiert nun in Zusammenarbeit mit KollegInnen des acib und der Universität Graz eine neue, auf künstlicher Intelligenz basierende Technologie, die die Gefährlichkeit von Virusvarianten zuverlässig klassifiziert. Dazu fließen globale und lokale Daten, wie zum Beispiel aus Abwasserproben, die rasch und umfassend auf ausgeschiedene Viren getestet werden, in die Berechnungen ein. Die Ergebnisse der Kooperation, die auf die Simulationsleistung von Amazon Web Services zurückgreift, sind kürzlich im Fachjournal „Scientific Reports“ aus dem Publikationshaus Nature erschienen.
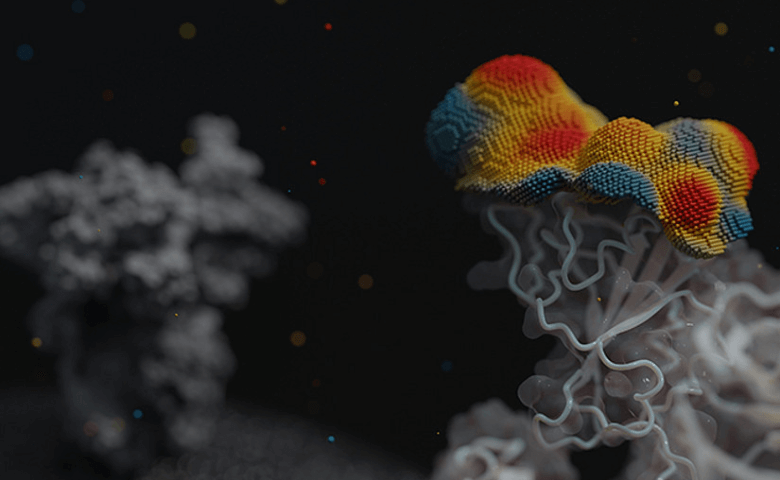
Bisherige Klassifizierungssysteme von Virusvarianten benötigten viel Zeit. Christian Gruber und sein Team nutzen dafür jetzt die von Innophore entwickelte Punktwolkentechnologie. Im ersten Schritt werden strukturelle Modelle der aktuellsten Virusvarianten erstellt. Aus diesen Modellen berechnen die ForscherInnen so genannte Punktwolken. „Diese geben uns Aufschluss darüber, wie stark die SARS-CoV-2-Spike-Rezeptorbindungsdomäne (RBD) mit dem menschlichen Rezeptor hACE2 interagiert. Das wiederum ist ein wichtiger Indikator für die Infektiosität“, erklärt Christian Gruber.
Ein wesentlicher Vorteil der Punktwolkentechnologie ist außerdem, dass die Methode auf künstlicher Intelligenz beruht und mit großen Datensätzen trainiert wird. Das erhöht die Genauigkeit und verbessert die Zuverlässigkeit der Vorhersagen. Das Einbinden von Simulationsleistung von Amazon Web Services erhöht die Geschwindigkeit der von Innophore durchgeführten Berechnungen zusätzlich um ein Vielfaches. Die Zusammenarbeit zwischen den beiden Unternehmen ist im Rahmen der Initiative zur Entwicklung von Diagnostika (DDI) entstanden und zu einer stabilen Kooperation gewachsen.
Publikation: Gruber et.al., Structural bioinformatics analysis of SARS-CoV-2 variants reveals higher hACE2 receptor binding affinity for Omicron B.1.1.529 spike RBD compared to wild type reference. Scientific Reports volume 12, Article number: 14534 (2022). https://doi.org/10.1038/s41598-022-18507-y